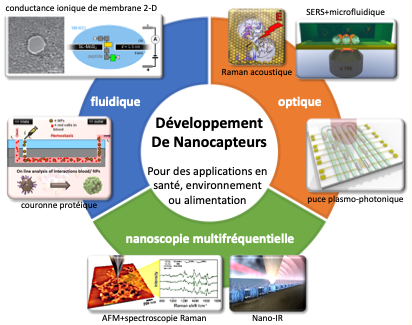
Développement de nanocapteurs et nanoscopie
Le département modélise et développe différents nanocapteurs et outils de nanocaractérisation innovants pour des applications dans les domaines de la santé, de l’environnement ou de l’alimentation.
- Développement de nanocapteurs plasmoniques/optiques
- Développement de nanocapteurs fluidiques
- Développement d’outils de caractérisation multifréquentielle à l’échelle nanométrique
- kc_data:
- a:8:{i:0;s:0:"";s:4:"mode";s:2:"kc";s:3:"css";s:0:"";s:9:"max_width";s:0:"";s:7:"classes";s:0:"";s:9:"thumbnail";s:0:"";s:9:"collapsed";s:0:"";s:9:"optimized";s:0:"";}
- kc_raw_content:
- [kc_row use_container="yes" _id="534476"][kc_column width="12/12" video_mute="no" _id="903913"][kc_spacing height="20" _id="67977"][/kc_column][/kc_row][kc_row use_container="yes" _id="135986"][kc_column width="60%" video_mute="no" _id="7789"][kc_title text="RMOpcGFydGVtZW50IE5hbm9zY2llbmNlcw==" _id="800495" type="h1" title_link="https://icb.u-bourgogne.fr/nanosciences/||"][kc_spacing height="40px" _id="706244"][kc_column_text _id="184898"]
Développement de nanocapteurs et nanoscopie
Le département modélise et développe différents nanocapteurs et outils de nanocaractérisation innovants pour des applications dans les domaines de la santé, de l’environnement ou de l’alimentation.
- Développement de nanocapteurs plasmoniques/optiques
{slide=Puces plasmo-photoniques pour la santé et l’agro-alimentaire}
En collaboration avec le département Photonique, nous développons des puces hybrides plasmo-photoniques fonctionnant dans le proche IR associées à la microfluidique appliquées au domaine agro-alimentaire. Ces puces intègrent des guides d’ondes, le plus souvent en configuration interférométrique avec des zones d’interaction plasmoniques avec le milieu biologique de dimensions micrométriques, permettant la détection de petites molécules (typiquement des contaminants tels que des pesticides, etc.) avec une sensibilité inégalée (objectif 25000 nm/RIU). L’intégration sur puce de nano-émetteurs (Q-dots) permettant d’optimiser et de réduire l’empreinte de ces capteurs sans difficultés d’alignement fait partie de nos voies de recherche, ainsi que le développement de modules microfluidiques couplés aux puces optiques permettant d’adresser le fluide localement sur les multiples zones de détection de la puce.
{/slide}
{slide=Pour la caractérisation de la conformation des protéines}De nombreuses pathologies sont identifiables à partir de marqueurs moléculaires (protéines) et la détection et caractérisation des structures de ces molécules biologiques nécessite le développement de capteurs, à l’échelle de la molécule unique. Les maladies neurodégénératives telles que Alzheimer et Parkinson sont caractérisées par l’observation à l’échelle moléculaire de dégâts irréversibles sur les neurones causées par des agrégats de protéines intrinsèquement désordonnées.
La caractérisation de ces protéines intrinsèquement désordonnées est un véritable challenge d’un point de vue théorique et expérimental. Le paysage d’énergie libre de ces systèmes présente de nombreux états métastables et peut être fortement déformé par l’interaction des protéines entre elles (oligomérisation) ou avec une protéine. Des approches de dynamique moléculaire tous atomes, à gros grains, et des modèles mathématiques issus de la théorie des graphes et de l’étude statistique de modèles sur réseaux (modèles HP, SAW) sont développés afin de caractériser la structure et la dynamique de ces protéines et de dégager des paramètres d’ordre mesurables par spectroscopies.
Des mesures expérimentales et des analyses fines des réponses spectroscopiques Raman, à l’échelle de la molécule unique, sont également réalisées afin d’accéder aux différents niveaux du désordre et de sonder les transitions de conformations régulées par des contraintes environnementales (température, potentiel électrochimique, pH, etc.). La démarche suppose d’intégrer plusieurs nanobriques : la plasmonique localisée pour augmenter la résolution spatiale, des spectroscopies à détection compressive pour accélérer la dynamique, de la microfluidique thermalisée et électroactive pour le contrôle de l’environnement, ainsi que des analyses statistiques graduées suivant la complexité du signal (Kmeans, PCA, diagramme de Markov, réseaux neuronaux).{/slide}
- Développement de nanocapteurs fluidiques
{slide=Pour l’étude de la couronne protéique}
L’étude de la couronne protéique adsorbée sur les nanoparticules est cruciale pour anticiper et améliorer les comportements biologiques des nanoparticules (NPs) notamment lorsqu’elles sont utilisées pour des applications biomédicales. En effet, une fois mises en contact avec des fluides biologiques, ces NPs vont se couvrir de protéines et cette adsorption va être influencée par la chimie de surface des matériaux développés mais va également influencer leurs devenirs biologiques. Ainsi, les mesures de protéines effectuées en statique in vitro sont très peu comparables à la réalité des mesures en conditions biologiques in vivo.
En collaboration avec le laboratoire FEMTO-ST, un micro-dispositif original permettant de simuler et caractériser la couronne protéique de nanoparticules en dynamique dans un flux simulant le flux sanguin sera élaboré. Il permettra ainsi de mieux comprendre sa formation et d’optimiser l’élaboration de nanoparticules pour des applications médicales tout en proposant des protocoles d’analyses standardisés de cette couronne protéique.
La couronne protéique sera également étudiée in vivo. Pour cela, le département nanosciences travaille avec les acteurs biologistes et médecins de la région (CHU, CGFL et INSERM) pour comprendre l’influence des protéines adsorbées à la surface des NPs sur leurs comportements biomédicaux (biodistribution, biocirculation et toxicité). Le but final de cette approche étant de développer des NPs biomimétiques fonctionnalisés possédant des propriétés biologiques plus circulantes et biocompatibles pour une nanomédecine personnalisée.{/slide}
{slide=Pour le séquençage de protéines}Le diagnostic rapide de maladies est un enjeu sanitaire majeur. De nombreuses pathologies sont identifiables à partir de marqueurs moléculaires (protéines) présents dans les fluides physiologiques (sang, salive, etc.). La détection biochimique des protéines nécessite en principe de développer un processus de détection ad hoc adapté à chaque marqueur. Au contraire, dans ce projet, nous étudions la détection de protéines par séquençage au moyen d’un dispositif universel issu des nanotechnologies appelé nanopore. Ce dispositif permet la détection de très faibles concentrations de protéines pour un diagnostic précoce. Le concept est basé sur la lecture physique de la carte d’identité de la protéine, c’est-à-dire de sa séquence d’acides aminés.
Le dispositif est constitué d'un nanopore percé dans une membrane de MoS2, plongée dans une solution ionique (typiquement KCl). La membrane est soumise à une différence de potentiel électrique, engendrant un courant ionique. Le principe est que ce courant ionique est bloqué/modifié lors du passage d’un biopolymère au travers du nanopore. La mesure ultra-rapide des fluctuations de courant fournit des informations structurales sur le passage des différents monomères de la biomolécule. L’analyse des chutes de courant et de leurs durées, interprétées comme des événements de traversées du pore permet en théorie d’identifier la nature de chaque monomère et ainsi d’en déduire la séquence de la protéine.
Afin d'extraire la séquence d'acides aminés traversant le nanopore et ainsi identifier la protéine marqueur de la maladie, la modélisation à l’échelle atomique du nanocapteur et la simulation par dynamique moléculaire, couplées à des techniques d’analyse et d’extraction de l’information de séries temporelles et de Machine Learning sont utilisées.{/slide}
- Développement d’outils de caractérisation multifréquentielle à l’échelle nanométrique
{slide=Interactions protéines/membranes biologiques}
Afin d’élucider d’un point de vue fondamental les mécanismes d’interactions entre les protéines de stress Lo18 (small HSP) et la membrane biologique, à l’échelle atomique, nous utilisons des nanosondes de type AFM capable de cartographier les systèmes à la fois de manière topographiques et spectrales (IR, Raman, micro-onde). L’identification des acide aminés impliqués dans l’activité lipochaperon est cruciale. Des études en microbiologie et microscope à force atomique ont montré que la dégradation de la membrane cellulaire par un agent chimique était stoppée par l’addition des protéines Lo18, présentes sous forme de dimère. Ce projet, pluridisciplinaire, consiste en la modélisation à l’échelle atomique des interactions de la protéine Lo18 et de ses mutants avec la membrane cellulaire au moyen de simulations de dynamique moléculaire. Le stress sur la membrane sera simulé par l’augmentation de la température. En parallèle, des études en microbiologie et la caractérisation des interactions par microscopie à force atomique sur l’effet de la température et des mutations seront réalisées pour élucider les mécanismes d’interactions.
{/slide}
{slide=Contrôle non destructif pour l’analyse de composants}L’optimisation de la durée de vie des produits évoluant dans des conditions de plus en plus sévères, dans un contexte de réduction des coûts, oblige les fabricants de composants à prendre en compte la notion de contraintes résiduelles mais aussi de défauts précoces (durabilité). Depuis 2015, le département Nansciences et l’entreprise ARDPI membres du Laboratoire mixte public/privé NanoSense Lab développent une solution unique et innovante dans le monde du contrôle non destructif, basée sur la microscopie de champ proche micro-onde (scanning microwave microscopy : SMM) Cette technique permet de réaliser la tomographie 2D et 3D des matériaux, avec une résolution nanométrique et une grande sensibilité.
{/slide}
{slide=Pour la pharmaceutique}Nous étudions la mise en forme des poudres pharmaceutiques par le procédé de compression en matrice. Grâce à une approche couplée expérience/simulation numérique, nous avons proposé une méthodologie innovante pour l’étude de ce procédé afin d’aboutir à une meilleure compréhension des phénomènes contribuant, au cours du procédé, au développement de la cohésion au sein du comprimé avec comme objectif final de mieux appréhender et prévoir les performances du produit final. L’étude, au niveau microscopique, de l’interaction entre les grains permet de comprendre, caractériser et modéliser correctement l’adhérence qui se crée entre les particules en prenant en compte les déformations sous contraintes au cours du cycle compression/décompression caractérisées à l’échelle macroscopique du comprimé. Le but est d’aboutir à des comparaisons réalistes entre essais expérimentaux et essais numériques, et ce autant à l’échelle des particules qu’à l’échelle d’un volume élémentaire représentatif du comprimé. L'élaboration d'un outil de modélisation de type « jumeau numérique » constituerait un avantage certain pour la compétitivité des fabricants de comprimés pharmaceutiques, dans un contexte économique et sociétal de réimplantation sur nos territoires des activités de fabrication des produits de santé.
{/slide}
{slide=Pour l’étude de l’assemblage et du remodelage de complexes nucléoprotéique}Nous développons de nouveaux outils d’imagerie moléculaire afin d’acquérir une vision dynamique de plusieurs étapes des mécanismes de recombinaison homologue (RH) et de la fusion non homologue (NHEJ). La RH est une voie complexe impliquée dans la réparation des ruptures de double brin (CDB) de l'ADN et le redémarrage des fourches de réplication bloquées. La RH est donc essentielle à l’intégrité génomique, mais des échecs dans ce processus hautement régulé peuvent également conduire à des arrêts de la réplication de l’ADN ou à une mauvaise ségrégation des chromosomes. La toxicité de la RH est à l’origine de diverses maladies, telles que l’anémie de Fanconi ou le syndrome de Bloom. Cependant, le NHEJ reste le mécanisme dominant par lequel les cellules de mammifère réparent les cassures double brin (CDB). Il est également impliqué dans le processus de réarrangement des gènes des immunoglobulines et des récepteurs des cellules T, appelé recombinaison VDJ.
Nous souhaitons analyser sur molécule unique les propriétés dynamiques de la protéine de réplication (RPA) par HS-AFM et tester leur influence sur l’assemblage et le désassemblage des protéines de la RH précoces sur l’ADN simple brin (ADNsb). Les premiers résultats obtenus par HS-AFM confirment la possibilité de voir RPA se déplacer le long de l’ADNsb (il faut savoir que cette expérience reste un challenge d’un point de vue expérimental). En outre, le mécanisme par lequel, sur un ADNsb recouvert de RPA, le médiateur Rad52 permet le chargement et la nucléation de Rad51 reste flou. L'étude envisagée de la formation et de la dynamique de Rad51 avec et sans Rad52 par HS-AFM s’attachera à clarifier si Rad52 est en mesure de rendre RPA plus mobile sur l'ADN simple brin, offrant ainsi une accessibilité à la liaison Rad51 ou bien si Rad52 se lie directement aux complexes RPA/ADNsb pour recruter Rad51.{/slide}
[/kc_column_text][kc_spacing height="10px" _id="137901"][/kc_column][kc_column width="10%" video_mute="no" _id="216303"][/kc_column][kc_column width="30%" video_mute="no" _id="708836"][kc_single_image image_size="full" _id="908376" image_source="media_library" image="42125"][kc_spacing height="40px" _id="139068"][kc_column_text _id="618309"]MEMBRES
[/kc_column_text][kc_spacing height="11px" _id="371816"][kc_single_image image_size="141x100" _id="501855" image_source="media_library" image="42334"][kc_column_text _id="672894"]{slide= Permanents}
Cyrille ANDRES (MCF)
Véronique BERARD (MCF)
Julien BOUDON (MCF)
Eric BOURILLOT (MCF)
Denis CHAUMONT (MCF)
Thierry DAVID (MCF)
Patrice DELARUE (MCF)
Alain DEREUX (PR)
Eric FINOT (PR)
Michaele HERBST (AI)
Françoise LEBRUN (T)
Aymeric LERAY (CR)
Eric LESNIEWSKA (PR)
Laurent MARKEY (IR)
Lionel MAURIZI (CR)
Nadine MILLOT (PR)
Adrien NICOLAI (MCF)
Olivier PIETREMENT (CR)
Lucien SAVIOT (DR)
Patrick SENET (PR){/slide}
{slide= Non-permanents}
Adem DAHI
Dorra BEN ELKADHI
Laura CONVERT
Irma CUSTOVIC
William D'ODET D'ORSONNENS
Marie GARNIER
Sylvain HERNANDEZ SABIO
Eduardo HERNANDO ABAD
Célia MARETS
Mélanie ROMAIN
Charif TAMIN
Arielle TCHINDA SONHAPI{/slide}
[/kc_column_text][kc_column_text _id="910756"]PUBLICATIONS
[/kc_column_text][kc_spacing height="11px" _id="261321"][kc_single_image image_size="85x100" _id="955865" image_source="media_library" image="42129" on_click_action="open_custom_link" custom_link="https://icb.u-bourgogne.fr/publications-scientifiques-nanosciences/|Publications scientifiques|"][/kc_column][/kc_row]
SCIENTIFIQUES