Des structures auto-assemblables de protéines artificielles ou comment élaborer un « origami » moléculaire
S’il est possible de construire des nanostructures de forme contrôlée en exploitant la structure en double hélice des molécules d’ADN (conduisant à des assemblages communément appelés « origamis d’ADN »), il demeure beaucoup plus difficile d’élaborer de telles structures précises à partir de protéines. Pourtant, dans les cellules vivantes, des architectures supramoléculaires très sophistiquées comme les microtubules, les filaments d’actine, ou les flagelles assurent des fonctions vitales et sont entièrement constituées de protéines naturelles. Ces dernières s’assemblent spontanément parce que chaque protéine a une forme particulière qui lui permet d’interagir de façon très spécifique avec d’autres protéines, menant ainsi à des architectures complexes. Créer des architectures supramoléculaires ordonnées de protéines ouvrirait la porte à de nombreuses applications en biologie mais aussi en science des matériaux.
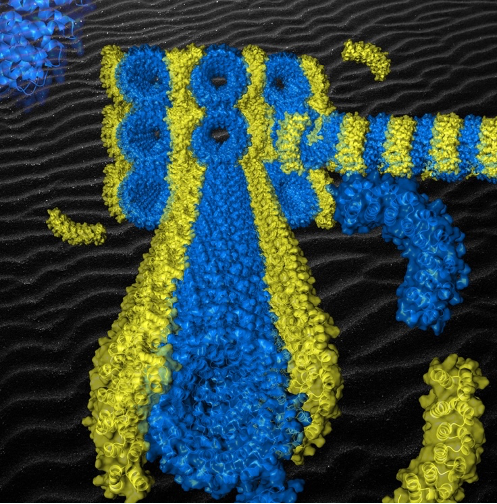
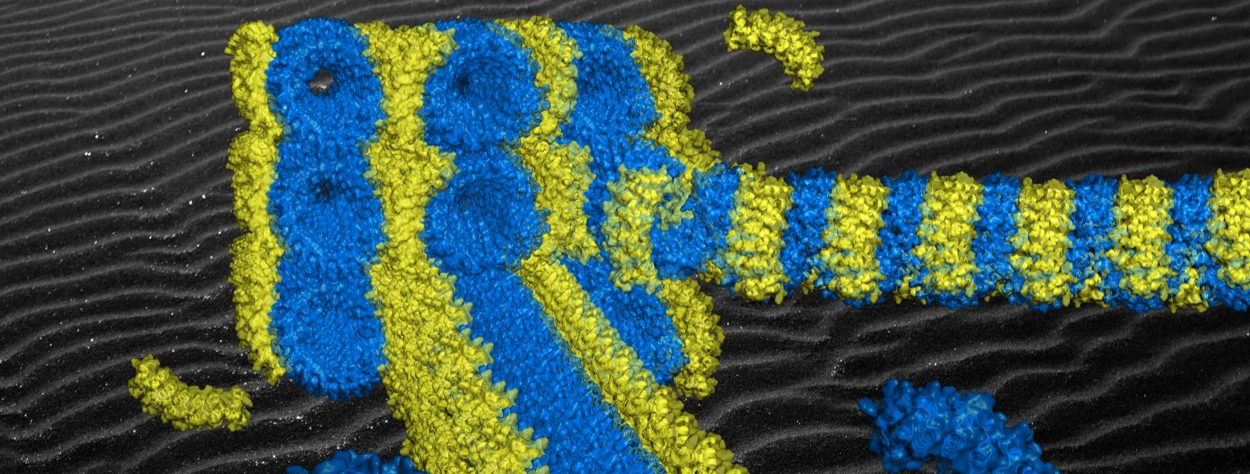
Figure : Modèle semi-expérimental de l'origami de protéines artificielles dont la superhélice de briques (en bleu) s'auto-assemble par affinité avec l'agrafe (en jaune). L'extrême régularité de la structure supramoléculaire obtenue dispose les agrafes périodiquement le long de la superhélice et induit la formation de cristaux d'origami alignés (voir au fond).
@ I2BC, CEMES, CBI, IPR et ICB
- Lire l'article de l'INP
- Design, synthesis, and characterization of protein origami based on self-assembly of a brick and staple artificial protein pair. L. Moreaud et al., PNAS, paru le 09 mars 2023
DOI : 10.1073/pnas.2218428120
Quatre équipes françaises démontrent la possibilité de construire des architectures supramoléculaires ordonnées qui se forment spontanément à partir de protéines spécialement conçues dans ce but. La clé de cette innovation est l'ingénierie de protéines très régulières, à motifs répétés, qui comportent des surfaces de reconnaissance leur permettant d’établir des interactions spécifiques.
Le Laboratoire Interdisciplinaire Carnot de Bourgogne (ICB), le Centre d'élaboration de matériaux et d'études structurales (CEMES, CNRS), l'Institut de biologie intégrative de la cellule ( I2BC, CNRS / CEA / Université Paris Saclay), l'Institut de physique de Rennes (IPR, CNRS / Université de Rennes), et le Centre de biologie intégrative (CNRS / Université de Toulouse – Paul Sabatier) ont élaboré méthode généralisable de construction d'architectures de protéines artificielles. Plutôt que de modifier des protéines naturelles, les scientifiques ont privilégié la conception de nouvelles protéines à la fois très régulières et programmées pour s'assembler dans des géométries précises capables de former des superstructures stables. L'une de ces protéines, dite « agrafe », a pour rôle d'agencer précisément plusieurs autres protéines appelées « briques », leur assemblage donnant naturellement à l'architecture tridimensionnelle supramoléculaire sa complexité structurale. Un premier défi a donc été de concevoir ces protéines et de les produire. Ensuite, en combinant plusieurs techniques dont la diffusion X et la cryomicroscopie électronique, les chercheurs ont montré ainsi que les protéines s'assemblent en quelques minutes, à température ambiante et suivant l'architecture prévue. Ceci constitue une première expérimentale dont le principe est généralisable à d’autres systèmes moléculaires.
Ces résultats sont publiés dans la revue Proceedings of the National Academy of Science.
- kc_data:
- a:8:{i:0;s:0:"";s:4:"mode";s:2:"kc";s:3:"css";s:0:"";s:9:"max_width";s:0:"";s:7:"classes";s:0:"";s:9:"thumbnail";s:0:"";s:9:"collapsed";s:0:"";s:9:"optimized";s:0:"";}
- kc_raw_content:
- [kc_row use_container="yes" _id="224688"][kc_column width="12/12" video_mute="no" _id="804256"][kc_spacing height="20" _id="227643"][kc_column_text _id="767988"]
S’il est possible de construire des nanostructures de forme contrôlée en exploitant la structure en double hélice des molécules d’ADN (conduisant à des assemblages communément appelés "origamis d'ADN"), il demeure beaucoup plus difficile d'élaborer de telles structures précises à partir de protéines. Pourtant, dans les cellules vivantes, des architectures supramoléculaires très sophistiquées comme les microtubules, les filaments d’actine, ou les flagelles assurent des fonctions vitales et sont entièrement constituées de protéines naturelles. Ces dernières s'assemblent spontanément parce que chaque protéine a une forme particulière qui lui permet d’interagir de façon très spécifique avec d’autres protéines, menant ainsi à des architectures complexes. Créer des architectures supramoléculaires ordonnées de protéines ouvrirait la porte à de nombreuses applications en biologie mais aussi en science des matériaux.
[/kc_column_text][kc_spacing height="20" _id="939959"][/kc_column][/kc_row][kc_row use_container="yes" _id="363345"][kc_column width="40%" _id="156413"][kc_spacing height="20" _id="477211"][kc_single_image image_size="full" _id="504714" image_source="media_library" image="42384"][kc_spacing height="6px" _id="918585"][kc_column_text _id="255565"]Figure : Modèle semi-expérimental de l'origami de protéines artificielles dont la superhélice de briques (en bleu) s'auto-assemble par affinité avec l'agrafe (en jaune). L'extrême régularité de la structure supramoléculaire obtenue dispose les agrafes périodiquement le long de la superhélice et induit la formation de cristaux d'origami alignés (voir au fond).
[/kc_column_text][kc_spacing height="10px" _id="142492"][kc_column_text _id="172638"]
@ I2BC, CEMES, CBI, IPR et ICB- Lire l'article de l'INP
- Design, synthesis, and characterization of protein origami based on self-assembly of a brick and staple artificial protein pair. L. Moreaud et al., PNAS, paru le 09 mars 2023
DOI : 10.1073/pnas.2218428120
Quatre équipes françaises démontrent la possibilité de construire des architectures supramoléculaires ordonnées qui se forment spontanément à partir de protéines spécialement conçues dans ce but. La clé de cette innovation est l'ingénierie de protéines très régulières, à motifs répétés, qui comportent des surfaces de reconnaissance leur permettant d’établir des interactions spécifiques.
[/kc_column_text][kc_column_text _id="287968"]Le Laboratoire Interdisciplinaire Carnot de Bourgogne (ICB), le Centre d'élaboration de matériaux et d'études structurales (CEMES, CNRS), l'Institut de biologie intégrative de la cellule ( I2BC, CNRS / CEA / Université Paris Saclay), l'Institut de physique de Rennes (IPR, CNRS / Université de Rennes), et le Centre de biologie intégrative (CNRS / Université de Toulouse - Paul Sabatier) ont élaboré méthode généralisable de construction d'architectures de protéines artificielles. Plutôt que de modifier des protéines naturelles, les scientifiques ont privilégié la conception de nouvelles protéines à la fois très régulières et programmées pour s'assembler dans des géométries précises capables de former des superstructures stables. L'une de ces protéines, dite "agrafe", a pour rôle d'agencer précisément plusieurs autres protéines appelées "briques", leur assemblage donnant naturellement à l'architecture tridimensionnelle supramoléculaire sa complexité structurale. Un premier défi a donc été de concevoir ces protéines et de les produire. Ensuite, en combinant plusieurs techniques dont la diffusion X et la cryomicroscopie électronique, les chercheurs ont montré ainsi que les protéines s'assemblent en quelques minutes, à température ambiante et suivant l'architecture prévue. Ceci constitue une première expérimentale dont le principe est généralisable à d’autres systèmes moléculaires.
[/kc_column_text][kc_column_text _id="754009"]Ces résultats sont publiés dans la revue Proceedings of the National Academy of Science.
[/kc_column_text][/kc_column][/kc_row]
